Vigilância da influenza aviária em aves silvestres utilizando diferentes técnicas diagnósticas na Costa Rica
Resumo
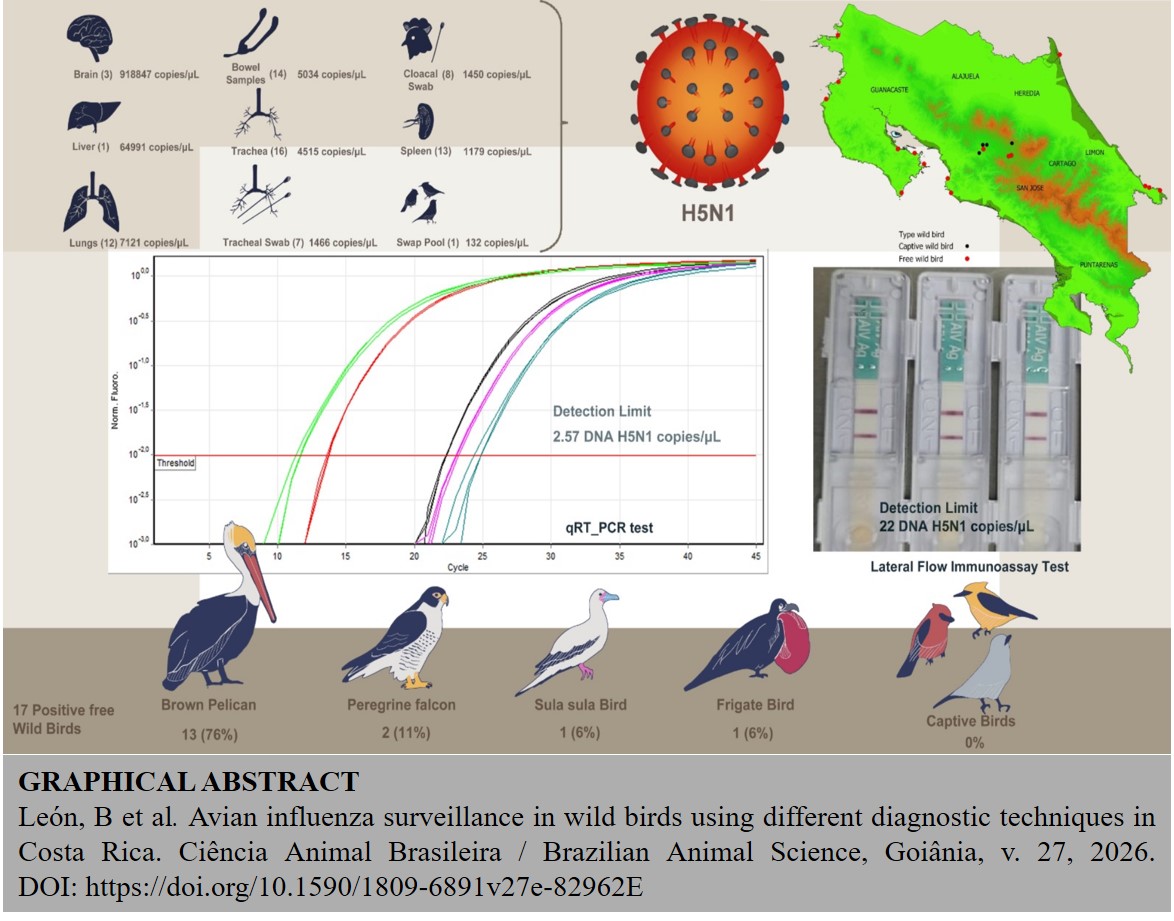
A vigilância do vírus da influenza aviária (VIA) é essencial para detectar precocemente surtos em aves silvestres. O presente estudo comparou a eficácia do teste rápido imunocromatográfico (TRI) com a PCR em tempo real (qRT-PCR). O TRI demonstrou maior sensibilidade que o teste de imunodifusão em gel de ágar (TIGA), detectando antígenos até a diluição 1:512, contra 1:16 do TIGA. Esses resultados foram confirmados por qRT-PCR, com valor médio de Ct de 34,5 (22 cópias de DNA viral/µL), e limite de detecção de 2,57 cópias/µL. Um total de 61 aves silvestres foram reportadas ao Serviço Nacional de Saúde Animal (SENASA) da Costa Rica entre janeiro e dezembro de 2023. Destas, 47 (77,04%) apresentaram sinais clínicos compatíveis com influenza aviária, enquanto 14 (22,96%) foram encontradas mortas. Foram identificadas treze ordens taxonômicas e 22 espécies de aves, sendo que 32,20 % das amostras pertenciam à ordem Pelecaniformes. Utilizando a técnica de qRT-PCR, 17 das 61 aves (27,86 %) testaram positivo para o vírus da influenza A (H5N1). Entre os casos positivos, 76,47 % eram Pelecanus occidentalis, 11,77 % Falco peregrinus, 5,88 % atobás-de-pé-vermelho (Sula sula) e 5,88 % fragatas-grandes (Fregata minor). Das 61 aves silvestres, 14 (7 positivas e 7 negativas) foram testadas tanto por RIT quanto por qRT-PCR, mostrando 100% de concordância entre os dois métodos. A análise geoespacial indicou maior concentração de casos na costa do Pacífico. A variação na carga viral entre diferentes tecidos reforça o risco de falsos negativos e a necessidade de otimização da coleta. Nenhuma das 200 amostras de suabes cloacais de aves em centros de resgate testou positivo por RIT. Os achados destacam a importância de métodos diagnósticos combinados e de vigilância contínua para o controle eficaz da influenza aviária na Costa Rica.
Palavras-chave: Zoonoses; aves; H5N1; aves silvestres.
Downloads
Referências
1. Hill NJ, Bishop MA, Trovão NS, Ineson KM, Schaefer AL, Puryear WB, et al. Ecological divergence of wild birds drives avian influenza spillover and global spread. PLoS Pathog. 2022;18:e1010062. Available at: https://doi.org/10.1371/journal.ppat.1010062
2. Venkatesh D, Poen MJ, Bestebroer TM, Scheuer RD, Vuong O, Chkhaidze M, et al. Avian influenza viruses in wild birds: virus evolution in a multihost ecosystem. J. Virol. Amer. Soc. Microbiol. 2018;92:10.1128/jvi.00433-18. Available at: https://doi.org/10.1128/jvi.00433-18
3. Gonzalez-Reiche AS, Müller ML, Ortiz L, Cordón-Rosales C, Perez DR. Prevalence and diversity of low pathogenicity avian influenza viruses in wild birds in Guatemala, 2010-2013. Avian Dis. 2016;60:359–364. Availble at: https://doi.org/10.1637/11130-050715-Reg
4. Fujimoto Y, Usui T, Ito H, Ono E, Ito T. Susceptibility of wild passerines to subtype H5N1 highly pathogenic avian influenza viruses. Avian Pathol. J, WVPA. 2015;44:243–247. Available at: https://doi.org/10.1080/03079457.2015.1043235
5. Munster VJ, Baas C, Lexmond P, Waldenström J, Wallensten A, Fransson T, et al. Spatial, temporal, and species variation in prevalence of influenza A viruses in wild migratory birds. PLoS Pathog. 2007;3:e61. Available at: https://doi.org/10.1371/journal.ppat.0030061
6. Knight-Jones TJD, Hauser R, Matthes D, Stärk KDC. Evaluation of effectiveness and efficiency of wild bird surveillance for avian influenza. Vet. Res. 2010;41:50. Available at: https://doi.org/10.1051/vetres/2010023
7. Perera CL, Díaz de Arce H, Pérez LJ. Actualización y perspectivas en el diagnóstico del virus de la influenza aviar. Rev. Salud Anim. 2011;33:01–7. Available at: https://www.produccion-animal.com.ar/produccion_aves/enfermedades_aves/112-influenza_actualizacion.pdf
8. Wibowo MH, Untari T, Artanto S, Putri K, Amanu S, Asmara W. Evaluation of rapid detection kit against avian influenza A virus and H5 subtype for field sample. Indones. J. Biotechnol. 2016;21:48–55. Available at: https://doi.org/10.22146/ijbiotech.26792
9. Spackman E, Senne DA, Myers TJ, Bulaga LL, Garber LP, Perdue ML, et al. Development of a real-time reverse transcriptase PCR assay for type A influenza virus and the avian H5 and H7 hemagglutinin subtypes. J. Clin. Microbiol. 2002;40:3256–3260. Available at: https://doi.org/10.1128/JCM.40.9.3256-3260.2002
10. León B, Chaves G, Koster LG, Jenkins-Moore M, Carrillo C, Méndez D. Isolation and identification of pandemic influenza virus H1N1 / 2009 S-OIV from commercial and backyard swine in Costa Rica. Rev. Cienc. Vet. 2011;29:53–81.
11. Bustin SA, Mueller R. Real-time reverse transcription PCR (qRT-PCR) and its potential use in clinical diagnosis. Clin. Sci. 2005;109:365–379. Available at: https://doi.org/10.1042/CS20050086
12. Yuan JS, Reed A, Chen F, Stewart CN. Statistical analysis of real-time PCR data. BMC Bioinform. 2006;7:85. https://doi.org/10.1186/1471-2105-7-85
13. Nassiri M, Torshizi, ME, Ghovvati S, Doosti M. Evaluation of different statistical methods using SAS software: an in silico approach for analysis of real-time PCR data. J. Appl. Stat. 2018;45:306–319. Available at: https://doi.org/10.1080/02664763.2016.1276890
14. Gall A, Hoffmann B, Harder T, Grund C, Beer M. Universal primer set for amplification and sequencing of HA0 cleavage sites of all influenza A viruses. J. Clin. Microbiol. 2008;46:2561–2567. Available at: https://doi.org/10.1128/JCM.00466-08
15. Zhou B, Donnelly ME, Scholes DT, George KS, Hatta M, Kawaoka Y, et al. Single-reaction genomic amplification accelerates sequencing and vaccine production for classical and swine origin human influenza A viruses. J. Virol. 2009;83:10309–10313. Available at: https://doi.org/10.1128/jvi.01109-09
16. Chua T-H, Ellis TM, Wong CW, Guan Y, Ge SX, Peng G, et al. Performance evaluation of five detection tests for avian influenza antigen with various avian samples. Avian Dis. 2007;51:96–105. Available at: https:// DOI.org/ 10.1637/0005-2086(2007)051[0096:PEOFDT]2.0.CO;2
17. Slomka MJ, Pavlidis T, Banks J, Shell W, McNally A, Essen S, et al. Validated H5 Eurasian real-time reverse transcriptase-polymerase chain reaction and its application in H5N1 outbreaks in 2005-2006. Avian Dis. 2007;51:373–377. Available at: https://doi.org/10.1637/7664-060906R1.1
18. Spackman E, Pedersen JC, McKinley ET, Gelb J. Optimal specimen collection and transport methods for the detection of avian influenza virus and Newcastle disease virus. BMC Vet. Res. 2013;9:35. Available at: https://doi.org/10.1186/1746-6148-9-35
19. McDuie F, T. Overton C, A. Lorenz A, L. Matchett E, L. Mott A, A. Mackell D, et al. Mitigating risk: predicting H5N1 avian influenza spread with an empirical model of bird movement. Transbound Emerg Dis. 2024;2024:5525298. Available at: https://doi.org/10.1155/2024/5525298
20. Lebarbenchon C, Sreevatsan S, Ramakrishnan MA, Poulson R, Goekjian V, Di Matteo JJ, et al. Influenza A viruses in American white pelican (Pelecanus erythrorhynchos). J. Wildl. Dis. 2010;46:1284–1289. Available at: https://doi.org/10.7589/0090-3558-46.4.1284
21. Adzic B, Goletic S, Pejoviс N, Vizi A, Yolshin N. First case of highly pathogenic avian influenza H5N1 in Montenegro. Acta Vet.-Beogr. 2024;74:145–158. Available at: https://doi.org/10.2478/acve-2024-0011
22. Alexandrou O, Malakou M, Catsadorakis G. The impact of avian influenza 2022 on Dalmatian pelicans was the worst ever wildlife disaster in Greece. Oryx. 2022;56:813–813. Available at: https://doi.org/10.1017/S0030605322001041
23. Stoimenov GM, Goujgoulova GV, Nikolov B, Petrova R, Teneva A, Dimitrova I. Histopathological findings in Dalmatian pelicans (Pelecanus crispus) naturally infected with avian influenza subtype A H5N1 in Bulgaria. J. Hell. Vet. Med. Soc. 2018;68:369–376. Available at: https://doi.org/10.12681/jhvms.15493
24. Cardona CJ, Xing Z, Sandrock CE, Davis CE. Avian influenza in birds and mammals. Comp. Immunol. Microbiol. Infect. Dis. 2009;32:255–273. Available at: https://doi.org/10.1016/j.cimid.2008.01.001
25. Lara Rivera DA. Determinación de anticuerpos circulantes contra el virus de influenza aviar en aves del Centro de Rehabilitación de Vida Silvestre ARCAS, Flores, Petén, Guatemala. [Thesis]: Guatemala: Universidad de San Carlos; 2008. Available at: http://www.repositorio.usac.edu.gt/7406/
26. Neu Toscano AE. Determinación de anticuerpos circulantes contra el virus de influenza aviar en aves del Parque Zoológico Minerva de Quetzaltenango, Guatemala. [Thesis]: Guatemala: Universidad de San Carlos; 2016. Available at: http://biblioteca.usac.edu.gt/tesis/10/10_1589.pdf
27. Slusher MJ, Wilcox BR, Lutrell MP, Poulson RL, Brown JD, Yabsley MJ, et al. Are passerinae bords reservoirs for influenza A viruses? J. Wildl. Dis. 2014;50:792–809. Available at: https://doi.org/10.7589/2014-02-043
28. Brown JD, Berghaus RD, Costa TP, Poulson R, Carter DL, Lebarbenchon C, et al. Intestinal excretion of a wild bird-origin H3N8 low pathogenic avian influenza virus in mallards (Anas platyrhynchos). J. Wildl. Dis. 2012;48:991–998. Available at: https://doi.org/10.7589/2011-09-280
29. Swayne DE, Slemons RD. Using mean infectious dose of high- and low-pathogenicity avian influenza viruses originating from wild duck and poultry as one measure of infectivity and adaptation to poultry. Avian Dis. 2008;52:455–460. Available at: https://doi.org/10.1637/8229-012508-Reg.1
Downloads
Arquivos adicionais
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2026 Ciência Animal Brasileira / Brazilian Animal Science

Este trabalho está licenciado sob uma licença Creative Commons Attribution 4.0 International License.
Autores que publicam nesta revista concordam com os seguintes termos:
- Autores mantém os direitos autorais e concedem à revista o direito de primeira publicação, com o trabalho simultaneamente licenciado sob a Licença Creative Commons Attribution que permite o compartilhamento do trabalho com reconhecimento da autoria e publicação inicial nesta revista.
- Autores têm autorização para assumir contratos adicionais separadamente, para distribuição não-exclusiva da versão do trabalho publicada nesta revista (ex.: publicar em repositório institucional ou como capítulo de livro), com reconhecimento de autoria e publicação inicial nesta revista.
- Autores têm permissão e são estimulados a publicar e distribuir seu trabalho online (ex.: em repositórios institucionais ou na sua página pessoal) a qualquer ponto antes ou durante o processo editorial, já que isso pode gerar alterações produtivas, bem como aumentar o impacto e a citação do trabalho publicado (Veja O Efeito do Acesso Livre).
Declaração de dados
-
Os dados de pesquisa estão contidos no próprio manuscrito


























