Genótipos de circovírus suíno Tipo 2 e PCV3 em amostras clínicas de suínos no Brasil
DOI:
https://doi.org/10.1590/1809-6891v25e-77826EResumo
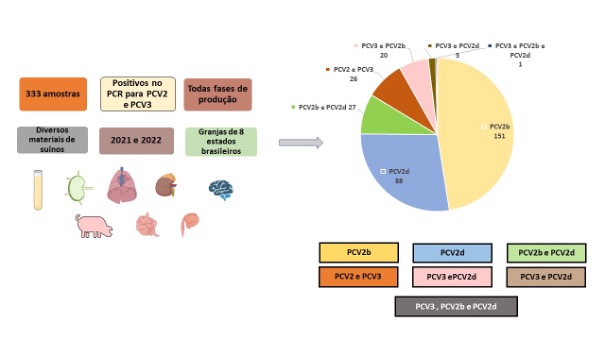
O Circovírus suíno tipo 2 (PCV2) é um importante patógeno para suínos e causador das doenças associadas a circovirose suína (PCVD). Apesar da PCVD ser controlada pelas vacinas comerciais, a sua apresentação está mudando. Além disso, o PCV2 está evoluindo geneticamente e novos genótipos foram identificados em suínos vacinados e não vacinados. O objetivo deste trabalho foi verificar a presença dos genótipos PCV2a, PCV2b e PCV2d em amostras previamente positivas para PCV2. Além disso, identificar coinfecções entre genótipos de PCV2 e/ou com PCV3, que também podem causar doenças em suínos, utilizando um teste de diagnóstico PCR rápido, eficaz e de baixo custo. Foram analisadas 333 amostras clínicas positivas para PCV2 por qPCR e provenientes de diferentes fases de produção e rebanhos do Brasil. Destas, 266 foram genotipadas, sendo o PCV2b o genótipo mais predominante (56,77% das amostras positivas), principalmente provenientes de animais da creche. O PCV2d também foi detectado em 33,10% das amostras, principalmente em suínos de terminação e porcas reprodutoras. O teste PCR utilizado neste estudo foi comparado a um kit comercial e foi eficaz na genotipagem do PCV2. Este estudo demonstrou a importância da genotipagem do PCV2 e que o PCV2b continua sendo o genótipo predominante responsável pela doença em granjas de suínos no Brasil. O PCV2a, genótipo mais comum utilizado nas vacinas comerciais, não foi detectado em nenhuma amostra analisada. Embora os suínos infectados com outros genótipos de PCV2 possam obter alguma proteção heteróloga das vacinas contra PCV2a, diagnósticos adequados e monitoria da vacina para atualizações devem ser considerados.
Downloads
Referências
Allan G, McNeilly F, Cassidy J, Reilly G, Adair B, Ellis W, et al. Pathogenesis of porcine circovirus; experimental infections of colostrum deprived piglets and examination of pig foetal material. Veterinary microbiology. 1995;44(1):49-64. https://doi.org/10.1016/0378-1135(94)00136-K
Ellis J, Hassard L, Clark E, Harding J, Allan G, Willson P, et al. Isolation of circovirus from lesions of pigs with postweaning multisystemic wasting syndrome. The Canadian veterinary journal. 1998;39(1):44. canvetj00147-0046.pdf (nih.gov)
Opriessnig T, Meng XJ, Halbur PG. Porcine circovirus type 2 associated disease: update on current terminology, clinical manifestations, pathogenesis, diagnosis, and intervention strategies. J Vet Diagn Invest. 2007;19(6):591-615. https://doi.org/10.1177/104063870701900601
Segalés J, Allan GM, Domingo M. Circoviruses. Diseases of swine. 2019:473-87. https://doi.org/10.1002/9781119350927.ch30
Ciacci-Zanella J, Mores N. Diagnosis of post-weaning multisystemic wasting syndrome in pigs in Brazil caused by porcine circovirus type 2. Arquivo Brasileiro De Medicina Veterinaria E Zootecnia. 2003;55(5):522-7. https://doi.org/10.1590/s0102-09352003000500002
Silva FMFd, Silva Júnior A, Peternelli EFdO, Viana VW, Chiarelli Neto O, Fietto JLR, et al. Retrospective study on Porcine circovirus-2 by nested pcr and real time pcr in archived tissues from 1978 in brazil. Brazilian Journal of Microbiology. 2011;42:1156-60. https://doi.org/10.1590/s1517-83822011000300039
Zanella JRC, Morés N, Barcellos DESNd. Main endemic health threats in the swine production chain in Brazil. Pesquisa Agropecuária Brasileira. 2016;51(5):443-53. https://doi.org/10.1590/s0100-204x2016000500004
Opriessnig T, Meng X-J, Halbur PG. Porcine circovirus type 2–associated disease: update on current terminology, clinical manifestations, pathogenesis, diagnosis, and intervention strategies. Journal of Veterinary Diagnostic Investigation. 2007;19(6):591-615. https://doi.org/10.1177/104063870701900601
Carman S, Cai HY, DeLay J, Youssef SA, McEwen BJ, Gagnon CA, et al. The emergence of a new strain of porcine circovirus-2 in Ontario and Quebec swine and its association with severe porcine circovirus associated disease—2004–2006. Canadian Journal of Veterinary Research. 2008;72(3):259. https://doi.org/10.1136/vr.167.7.260
Xiao CT, Halbur PG, Opriessnig T. Complete genome sequence of a novel porcine circovirus type 2b variant present in cases of vaccine failures in the United States. J Virol. 2012;86(22):12469. https://doi.org/10.1128/jvi.02345-12
Yao J, Qin Y, Zeng Y, Ouyang K, Chen Y, Huang W, et al. Genetic analysis of porcine circovirus type 2 (PCV2) strains between 2002 and 2016 reveals PCV2 mutant predominating in porcine population in Guangxi, China. BMC veterinary research. 2019;15:1-11. https://doi.org/10.1186/s12917-019-1859-z
Opriessnig T, O’Neill K, Gerber PF, de Castro AM, Gimenéz-Lirola LG, Beach NM, et al. A PCV2 vaccine based on genotype 2b is more effective than a 2a-based vaccine to protect against PCV2b or combined PCV2a/2b viremia in pigs with concurrent PCV2, PRRSV and PPV infection. Vaccine. 2013;31(3):487-94. https://doi.org/10.1016/j.vaccine.2012.11.030
Gava D, Serrao VHB, Fernandes LT, Cantao ME, Ciacci-Zanella JR, Mores N, et al. Structure analysis of capsid protein of Porcine circovirus type 2 from pigs with systemic disease. Braz J Microbiol. 2018;49(2):351-7. https://doi.org/10.1016/j.bjm.2017.08.007
Franzo G, Segalés J. Porcine circovirus 2 (PCV-2) genotype update and proposal of a new genotyping methodology. PLoS One. 2018;13(12):e0208585. https://doi.org/10.1371/journal.pone.0208585
Lv Q, Wang T, Deng J, Chen Y, Yan Q, Wang D, et al. Genomic analysis of porcine circovirus type 2 from southern China. Vet Med Sci. 2020;6(4):875-89. https://doi.org/10.1002/vms3.288
Wang Y, Noll L, Lu N, Porter E, Stoy C, Zheng W, et al. Genetic diversity and prevalence of porcine circovirus type 3 (PCV3) and type 2 (PCV2) in the Midwest of the USA during 2016-2018. Transbound Emerg Dis. 2020;67(3):1284-94. https://doi.org/10.1111/tbed.13467
Opriessnig T, Karuppannan AK, Castro AM, Xiao C-T. Porcine circoviruses: Current status, knowledge gaps and challenges. Virus research. 2020;286:198044. https://doi.org/10.1016/j.virusres.2020.198044
Assao VS, Santos MR, Rosado NCL, Bressan GC, Fietto JLR, Chang YF, et al. Genetic diversity of porcine circovirus 3 strains and the first detection of two different PCV3 strains coinfecting the same host in Minas Gerais, Brazil. Arch Virol. 2021;166(5):1463-8. https://doi.org/10.1007/s00705-021-05032-y
Rodrigues ILF, Cruz ACM, Souza AE, Knackfuss FB, Costa CHC, Silveira RL, et al. Retrospective study of porcine circovirus 3 (PCV3) in swine tissue from Brazil (1967-2018). Braz J Microbiol. 2020;51(3):1391-7. https://doi.org/10.1007/s42770-020-00281-6
ABPA. Brazilian Association of Animal Protein - Annual Report - 2023. 2023 [Available from: https://abpa-br.org/wp-content/uploads/2023/04/ABPA.-Annual-Report-2023..pdf.
EMBRAPA. CIAS - Central de Inteligencia de Aves e Suinos Brazil: Embrapa Swine and Poultry; 2023 [Available from: https://www.embrapa.br/en/suinos-e-aves/cias/estatisticas.
CEDISA. Centro de Diagnostico em Sanidade Animal 2023 [Available from: http://www.cedisa.org.br/home/.
Sibila M, Rocco C, Franzo G, Huerta E, Domingo M, Nunez JI, et al. Genotyping of Porcine Circovirus 2 (PCV-2) in Vaccinated Pigs Suffering from PCV-2-Systemic Disease between 2009 and 2020 in Spain. Pathogens. 2021;10(8). https://doi.org/10.3390/pathogens10081016
Dupont K, Nielsen E, Baekbo P, Larsen L. Genomic analysis of PCV2 isolates from Danish archives and a current PMWS case–control study supports a shift in genotypes with time. Veterinary microbiology. 2008;128(1-2):56-64. https://doi.org/10.1016/j.vetmic.2007.09.016
Hesse R, Kerrigan M, Rowland RR. Evidence for recombination between PCV2a and PCV2b in the field. Virus research. 2008;132(1-2):201-7. https://doi.org/10.1016/j.virusres.2007.10.013
Kim S-C, Nazki S, Kwon S, Juhng J-H, Mun K-H, Jeon D-Y, et al. The prevalence and genetic characteristics of porcine circovirus type 2 and 3 in Korea. BMC veterinary research. 2018;14:1-9. https://doi.org/10.1186/s12917-018-1614-x
Kwon T, Lee D-U, Yoo SJ, Sang HJ, Shin JY, Lyoo YS. Genotypic diversity of porcine circovirus type 2 (PCV2) and genotype shift to PCV2d in Korean pig population. Virus Research. 2017;228:24-9. https://doi.org/10.1016/j.virusres.2016.11.015
Mone NK, Clark NJ, Kyaw-Tanner M, Turni C, Barnes TS, Parke CR, et al. Genetic analysis of porcine circovirus type 2 (PCV2) in Queensland, Australia. Aust Vet J. 2020;98(8):388-95. https://doi.org/10.1111/avj.12952
Gagnon CA, Tremblay D, Tijssen P, Venne M-H, Houde A, Elahi SM. The emergence of porcine circovirus 2b genotype (PCV-2b) in swine in Canada. The Canadian Veterinary Journal. 2007;48(8):811. https://doi.org/10.1016/j.vetmic.2009.09.072
Franzo G, Segales J. Porcine Circovirus 2 Genotypes, Immunity and Vaccines: Multiple Genotypes but One Single Serotype. Pathogens. 2020;9(12). https://doi.org/10.3390/pathogens9121049
Ouyang T, Zhang X, Liu X, Ren L. Co-infection of swine with porcine circovirus type 2 and other swine viruses. Viruses. 2019;11(2):185. https://doi.org/10.3390/v11020185
Park KH, Chae C. The prevalence of porcine circovirus type 2e (PCV2e) in Korean slaughter pig lymph nodes when compared with other PCV2 genotypes. Transbound Emerg Dis. 2021;68(6):3043-7. https://doi.org/10.1111/tbed.13975
Downloads
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2024 Ciência Animal Brasileira / Brazilian Animal Science

Este trabalho está licenciado sob uma licença Creative Commons Attribution 4.0 International License.
Autores que publicam nesta revista concordam com os seguintes termos:
- Autores mantém os direitos autorais e concedem à revista o direito de primeira publicação, com o trabalho simultaneamente licenciado sob a Licença Creative Commons Attribution que permite o compartilhamento do trabalho com reconhecimento da autoria e publicação inicial nesta revista.
- Autores têm autorização para assumir contratos adicionais separadamente, para distribuição não-exclusiva da versão do trabalho publicada nesta revista (ex.: publicar em repositório institucional ou como capítulo de livro), com reconhecimento de autoria e publicação inicial nesta revista.
- Autores têm permissão e são estimulados a publicar e distribuir seu trabalho online (ex.: em repositórios institucionais ou na sua página pessoal) a qualquer ponto antes ou durante o processo editorial, já que isso pode gerar alterações produtivas, bem como aumentar o impacto e a citação do trabalho publicado (Veja O Efeito do Acesso Livre).






























