DOI: 10.1590/1089-6891v18e-38048
ZOOTECNIA
ESTRUTURA POPULACIONAL DO REBANHO NELORE CRIADO NO SEMIÁRIDO DO NORDESTE BRASILEIRO
POPULATION STRUCTURE OF THE NELLORE HERD REARED IN THE BRAZILIAN NORTHEAST SEMI-ARID
Marcos Paulo Gonçalves Rezende1*
Emanoela Aragão Souza Lisboa Conde1
Ana Carla Borges1
Paulo Luiz Souza Carneiro1
Raimundo Martins Filho2
Carlos Henrique Mendes Malhado1
1Universidade Estadual do Sudoeste da Bahia, Jequié, BA, Brasil
2Universidade Federal do Ceara, Fortaleza, CE, Brasil
* Autor para correspondência – mpgrezende@gmail.com
Resumo
Objetivou-se avaliar a estrutura populacional de 91.579 animais da raça Nelore criados no Semiárido Nordestino, nascidos entre 1965 e 2011. Analisaram-se o pedigree e a estimação dos parâmetros populacionais baseados na probabilidade de origem do gene. Nos primeiros anos, registraram-se nascimentos superiores de fêmeas, tendendo ao equilíbrio entre os sexos com o passar dos anos. Os números de efetivo de animais fundadores (fe) e ancestrais (fa) indicaram a utilização reduzida de animais na formação genética do rebanho. Dentre 24.676 ancestrais, 450 foram responsáveis por 50% da variabilidade genética da população. Dos 10 fundadores de maior importância, 8 se apresentam dentre os 10 ancestrais; o indivíduo com maior expressão apresentou coeficiente de relação médio (CR) de 1,31%, explicando 1,81% da variabilidade. 72% dos animais têm pais e mães identificados, observando-se perda de informação entre as gerações. 100% dos rebanhos utilizam touros externos e 61% deles vendem touros, não havendo classificação núcleo ou isolado. Endogamia variou de 0,14% na segunda geração a 0,73% na oitava, enquanto que o CR oscilou de 0,8% a 0,35% entre a primeira e a quarta geração, decrescendo a partir da quinta. Observou-se um intervalo médio de gerações de 8,3 anos. O rebanho possui número reduzido de animais na formação genética e pequena integralidade do pedigree, dificultando a estimativa de alguns parâmetros populacionais.
Palavras-chave: endogamia; intervalo de gerações; variabilidade genética; zebuínos.
Abstract
The objective of this study was to evaluate the population structure of 91,579 Nellore cattle reared in the Northeast semi-arid, born between 1965 and 2011. We analyzed the pedigree and the estimation of population parameters based on the probability of gene origin. In the early years, a higher number of female births occured, tending to gender balance over the years. The number of founder animals (fe) and ancestors (fa) indicated the reduced use of animals in the genetic formation of the herd. Among 2,4676 ancestors, 450 were responsible for 50% of the genetic variability of the population. Of the 10 most important founders, 8 were among the 10 ancestors. The individual with the highest expression presented a mean coefficient of relationship (CR) of 1.31%, explaining 1.81% of the variability.72% of animals have identified fathers and mothers, revealing loss of information between generations. 100% of the herds use external bulls and 61% of them sell bulls, with no core classification or isolate. Inbreeding ranged from 0.14% in the second generation to 0.73% in the eighth, while the CR ranged from 0.8% to 0.35% between the first and fourth generations, decreasing from the fifth on. The average generation interval observed was of 8.3 years. The herd has a small number of animals in the genetic make-up, and small completeness of pedigree, making it difficult to estimate some population parameters.
Keywords: generation interval; genetic variability; inbreeding; Zebu.
Recebido em: 14 outubro de 2015
Aceito em: 25 julho de 2017
Introdução
As projeções, em milhões de toneladas, para produção, consumo e exportação de carne bovina entre os anos de 2010 a 2020 indicam aumento de 16,75%, 17,13% e 14,55%, respectivamente(1), e o produtor deve adotar tecnologias em seu sistema de produção para torna-se competitivo nesse mercado.
Uma das ferramentas para elevar a produtividade do rebanho é a seleção(2,3), na qual se prioriza o acasalamento de indivíduos geneticamente superiores, de modo que as gerações futuras sejam mais eficientes, quando comparadas à geração atual. Adicionalmente, o conhecimento da estrutura da população e da variabilidade intrapopulacional por meio do pedigree pode nortear programas de seleção mais eficazes, orientando a gestão das estratégias de acasalamentos(4). Neste sentido, diversos autores(4-6) recomendaram evitar acasalamentos endogâmicos e inspecionar os acasalamentos, com base no parentesco entre os indivíduos, visando manter as taxas de endogamia baixas.
Metodologias para análises do pedigree e estimação dos parâmetros populacionais baseados na probabilidade de origem do gene, endogamia, intervalo de gerações, tamanho efetivo, coeficiente de relação médio, integralidade do pedigree, número de gerações e classificação dos rebanhos de acordo com a origem e uso dos touros estão descritas na literatura(7-9). Todavia, a literatura não relata estudos avaliando a estrutura populacional de rebanhos bovinos no Semiárido nordestino brasileiro.
Produzir bovinos nesse bioma é um desafio, devido às condições climáticas, com temperaturas variando de 23 à 42 ºC, umidade do ar baixa, regime de chuvas irregular e longos períodos de escassez de alimento e água(10). Portanto, fornecer informações quanto à estrutura populacional de bovinos Nelore criados nessa sub-região torna-se fundamental para auxiliar o pecuarista no melhoramento genético de seu rebanho.
Objetivou-se avaliar a estrutura populacional do rebanho de animais da raça Nelore criados no Semiárido do Nordeste Brasileiro nascidos entre os anos de 1965 a 2011.
Material e Métodos
Os dados são provenientes do Controle de Desenvolvimento Ponderal da Associação Brasileira de Criadores de Zebu (ABCZ). Utilizaram-se informações do pedigree de 91.579 animais (machos = 35.664 e fêmeas = 55.915), pertencentes a um rebanho de bovinos Nelore nascidos entre os anos de 1965 a 2011 na região Semi-Arida do Brasil, que engloba 92,97% do território do Rio Grande do Norte, 87,60% de Pernambuco, 86,74% do Ceará, 86,20% da Paraíba, 69,31% da Bahia, 59,41% do Piauí, 50,67% de Sergipe e 45,28% de Alagoas(11).
As análises foram realizadas utilizando-se o software ENDOG(12). A estrutura do pedigree foi obtida conforme sugerido por MacCluer et al.(13). O número de gerações completas traçadas (g), o número máximo de gerações traçadas e o número equivalente de gerações completas foram definidos como a geração mais distante em que todos os ancestrais (2g) do indivíduo são conhecidos; indivíduos com pais desconhecidos foram considerados fundadores (geração 0); o equivalente foi obtido por meio do somatório sobre todos os ancestrais conhecidos (1/2)n, em que n é o número de gerações que separa o indivíduo de cada ancestral conhecido(7).
Para o cálculo do coeficiente de endogamia (F), utilizou-se o algoritmo proposto por Meuwissen & Luo(8). O incremento de endogamia (ΔF), calculado para cada geração, foi obtido por: 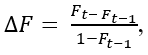 com rearranjos
com rearranjos 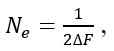 , em que: Ft = coeficiente médio de endogamia estimado na geração atual; Ft-1= coeficiente médio de endogamia estimado na geração anterior.
, em que: Ft = coeficiente médio de endogamia estimado na geração atual; Ft-1= coeficiente médio de endogamia estimado na geração anterior.
O Ne foi estimado baseado na variação de tamanho da família como:
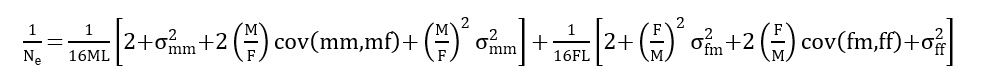
Em que M e F são o número de indivíduos masculinos e femininos, respectivamente, nascidos ou amostrados na reprodução para cada período de tempo, L é o intervalo médio de geração, σ²mm e σ²mf são as variâncias da prole masculina e feminina de um macho, σ²fm e σ²ff são as variâncias do sexo masculino e feminino da prole de uma fêmea, e cov (mm, mf) e cov (fm, ff) das respectivas covariâncias(14).
O coeficiente de relação médio (CR) foi definido como a probabilidade que um alelo escolhido aleatoriamente a partir de toda a população da linhagem pertença a um determinado animal. Utilizou-se um algoritmo para se obter um vetor c' definido como: c' = (1/n) 1'A [1]; A: matriz de relações sendo o numerador de tamanho n x n. Por outro lado, o numerador matriz de relações pode ser obtido a partir da matriz P, em que pij é igual a 1 se j é pai de i, e 0, caso contrário, que define os pais dos animais(15).
O número efetivo de fundadores (fe) foi dado por: 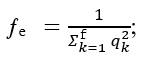 em que: ƒe = número efetivo de fundadores; qk = probabilidade do alelo ser originado do fundador k(9). O número efetivo de ancestrais (ƒa) foi determinado computando-se a contribuição marginal de cada ancestral por:
em que: ƒe = número efetivo de fundadores; qk = probabilidade do alelo ser originado do fundador k(9). O número efetivo de ancestrais (ƒa) foi determinado computando-se a contribuição marginal de cada ancestral por: 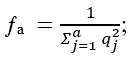 em que: ƒa = número efetivo de ancestrais; qj = contribuição marginal de um ancestral j (não necessariamente fundador), ou seja, a contribuição genética de ancestral que não é explicada por um ancestral escolhido anteriormente.
em que: ƒa = número efetivo de ancestrais; qj = contribuição marginal de um ancestral j (não necessariamente fundador), ou seja, a contribuição genética de ancestral que não é explicada por um ancestral escolhido anteriormente.
Definiu-se o intervalo de gerações (IEG) pela idade média dos pais no nascimento de sua progênie mantida para a reprodução ou não(16), calculado por meio de quatro passagens gaméticas (pai-filho, pai-filha, mãe-filho e mãe-filha), utilizando-se as datas de nascimento de cada indivíduo e dos seus pais, de acordo a seguinte fórmula: L = ¼ (LPM + LPF + LMM + LMF); em que: LPM = intervalo médio entre pais e filhos; LPF = intervalo médio entre pais e filhas; LMM = intervalo médio entre mães e filhos; LMF = intervalo médio entre mães e filhas.
Resultados e Discussão
Nos primeiros anos, houve superioridade nos registros dos nascimentos de fêmeas, tendendo a um equilíbrio entre os sexos com passar dos anos (Figura 1). Todavia, em todo o período, as fêmeas obtiveram quase 20% de registros a mais que os machos e, como o esperado é 50% de cada sexo, provavelmente, alguns machos podem não ter sido registrados.
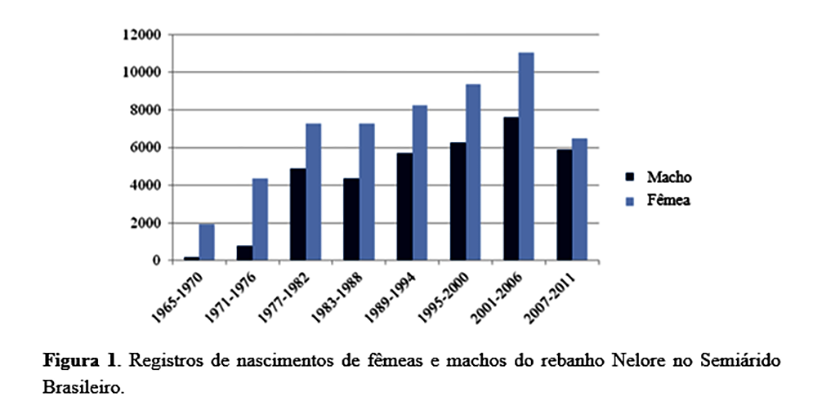
Esse fato pode ocasionar acasalamentos de animais aparentados, principalmente nos primeiros anos, pois, para que um animal seja registrado, ambos os pais devem ser registrados e esse menor registro de machos pode ter limitado a utilização de touros, alterando a frequência genotípica, mas não as frequências gênicas, acarretando a endogamia(6, 17).
O número efetivo de animais fundadores (fe = 959,35) foi superior ao número efetivo de ancestrais (fa = 482) (Tabela 1), sugerindo-se que houve contribuição de forma efetiva para composição genética do rebanho de animais além dos considerados fundadores. O ideal é que o número de animais efetivos sejam os mais próximos possíveis do tamanho da população fundadora(18).

A razão fe/fa foi de 1,99, indicando a utilização de um pequeno número de animais na formação genética do rebanho, caracterizando o efeito fundador, resultando em redução da variabilidade genética. Vozzi et al.(19) analisaram a raça Nelore, Muniz et al.(20) a raça Gir Mocha e Canaza-Cayo et al.(21) a raça Girolando e verificaram menor efeito fundador em relação ao verificado no presente estudo.
Dentre os 24.676 ancestrais, 450 foram responsáveis por 50% da variabilidade genética da população. Valores inferiores foram relatados na região Nordeste para raças Gir Mocha(20) e Indubrasil(22) com 39 e 225 ancestrais, respectivamente, explicando 50% da variabilidade genética da população. De acordo com Faria et al.(23), a preservação da variabilidade genética em determinada população é fundamental para elevar o retorno a curto e longo prazo da seleção, bem como para fins de conservação.
Dos dez fundadores de maior importância, oito se apresentam dentre os dentre ancestrais de maior contribuição (Tabela 2). O fundador e ancestral com as maiores contribuições foi o 1353767, macho, com coeficiente de relação médio (CR) de 1,31%, explicando 1,81% da variabilidade para a raça. Esses valores são inferiores aos verificados por Muniz et al.(20), que encontraram valores de CR de 35,25%, explicando 8,26% da variabilidade.
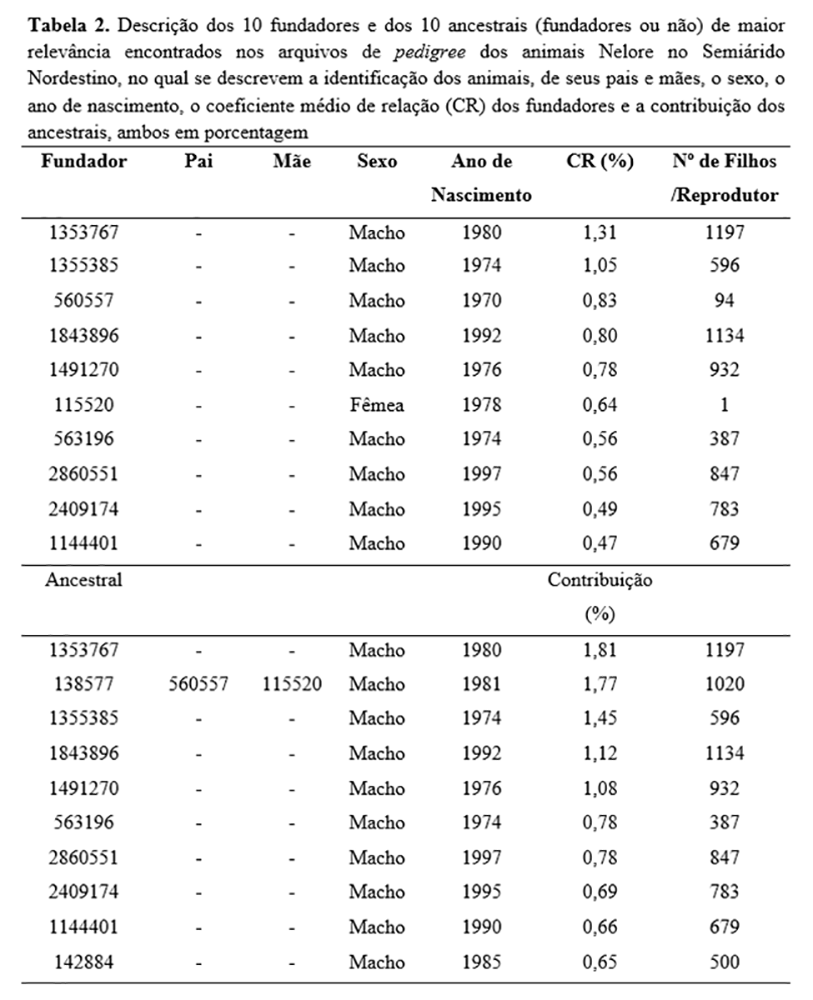
Dentre os 91.579 animais, 72,21% possuem mães identificadas e 72,29% pais identificados. Para a linha paterna, as informações são menos completas em relação à linha materna, corroborando o que foi apresentado na Figura 1. Foi observado que 72,25%; 20,74%; 5,98% e 1,46% apresentaram ascendência conhecida na primeira, segunda, terceira e quarta geração, respectivamente. Isso demonstra expressiva perda de informação entre a primeira e a segunda geração, pois a ausência de genealogia na primeira foi de 27,75% e na segunda de 79,2%. A elevação do aumento de gerações conhecidas possibilita estimativas mais acuradas sobre a estrutura das populações, fundamental para procedimentos de melhoramento genético da raça(4, 22).
Quando não há informações sobre a estrutura da população e mediante a ocorrência de acasalamentos entre animais oriundos de um mesmo rebanho (linhagem genética estreita), pode ocorrer aumento no grau de parentesco entre os indivíduos, elevando a endogamia(6).
A baixa integralidade do pedigree se dá pelo fato de que os registros genealógicos, mesmo tendo sido introduzidos no Brasil em 1919, só foi intensificado a partir de 1938, com as padronizações raciais das raças Gir, Nelore, Guzerá e Indubrasil, mas ainda se registravam animais com genealogia desconhecida de um ou ambos ascendentes(24). Apenas por volta do ano de 1971, a ABCZ determinou o que se definiu como "Livro Fechado", no qual foram inscritos todos os zebuínos registrados até aquela data e seus descendentes, não permitindo mais a entrada de animais com genealogia desconhecida(24).
Tal atitude favorece o aporte genético do rebanho, pois com o conhecimento genealógico conciso possibilita melhor ordenamento pelos programas de melhoramento, possibilitando estimativas de parâmetros populacionais mais confiáveis daqui algumas décadas.
O pequeno número de ancestrais conhecidos é corroborado pelo baixo número médio de gerações, demonstrando probabilidade de incremento na endogamia ao se conhecer maior número de gerações (Tabela 3).
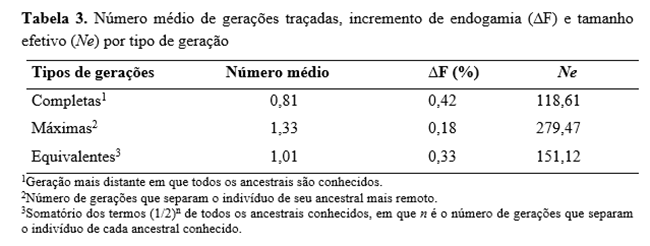
Nas gerações máximas (ancestrais mais remotos), o número médio e o ΔF associado ao maior Ne das três gerações confirmam que são inversamente proporcionais. Para geração equivalente, que considera todos os ancestrais, observaram-se valores medianos para o número médio, o ΔF e o Ne. Portanto, quando o pedigree é raso, subestima-se a endogamia e superestima-se o Ne.
Verificou-se que 100% dos rebanhos utilizam touros externos e que 61% deles vendem touros (Tabela 4). Classificou-se o rebanho registrado como 61,5% multiplicadores e 38,5% comercial. Não houve classificação como núcleo ou sem ligação (isolado). Vale destacar que é fundamental a migração e fluxo gênico entre os rebanhos, de modo a evitar subdivisão, agravando os problemas de conservação de grupos genéticos(22).

Na avaliação dos parâmetros endogâmicos, observou-se que o coeficiente de endogamia médio (F) sofreu incremento gradativo até a sexta geração (Tabela 5); a mesma tendência ocorreu com a porcentagem de indivíduos endogâmicos. O coeficiente de endogamia médio (F) variou de 0,14% na segunda geração a 0,73% na oitava, atingindo pico máximo na sexta geração (1,35%). Portanto, quanto maior a profundidade do pedigree, melhor precisão no cálculo da endogamia.
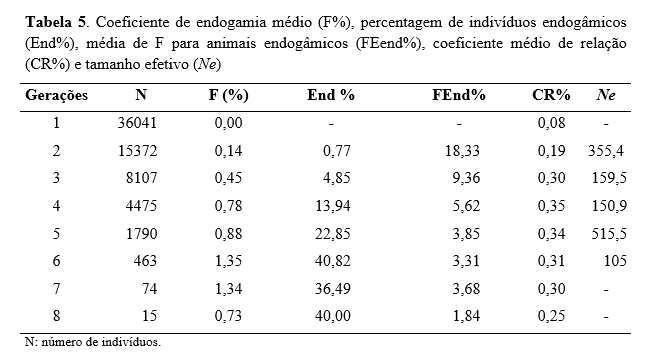
Efeitos desvantajosos do processo de endogamia nos rebanhos vêm sendo apresentados na literatura em relação à habilidade reprodutiva de vacas e novilhas associadas ao valor adaptativo(25), como também à taxa de crescimento e consequentemente ao desempenho dos animais(6).
Carvalheiro e Pimentel(26) reforçam que um motivo que justifica a utilização da endogamia é garantir a uniformidade racial (padronizações) e fixação de característica intrínseca a determinadas linhagens de touros, sendo praticada comumente por criadores que possuem animais registrados (raças puras). Todo esse processo foi intensificado após o desenvolvimento de biotecnologias reprodutivas, tornando os rebanhos mais favoráveis ao incremento da endogamia(21).
O coeficiente de relação médio (CR) apresentou valores de 0,8% a 0,35% entre a primeira e a quarta geração, voltando a decrescer a partir da quinta geração. O coeficiente de relação médio (CR) de cada indivíduo define-se como a probabilidade de um alelo escolhido ao acaso na população pertencer a determinado animal(27).
Barros et al.(18) relataram que uma maneira mais eficiente para o controle da endogamia a longo prazo é dar preferência ao uso de reprodutores com valores de coeficiente de relação médio (CR) baixos. Para Goyache et al.(28), baixo coeficiente de relação médio (CR) representa animais que compartilham porcentagem pequena de alelos com a população. Essa hipótese pode ser reforçada considerando os relatos de Gutiérrez & Goyache(12) de que o coeficiente de relação médio (CR) pode ser interpretado como a representação do animal em todo o pedigree; portanto, ele pode ser utilizado como complemento ao coeficiente de endogamia.
O valor médio da endogamia foi de 0,05% a partir de 1982 (0,8 gerações equivalentes), elevando-se rapidamente para 0,16% no ano de 1985 (Figura 2). Nos anos seguintes, os valores oscilaram até atingir 0,36% em 1995 (1,17 gerações equivalentes).
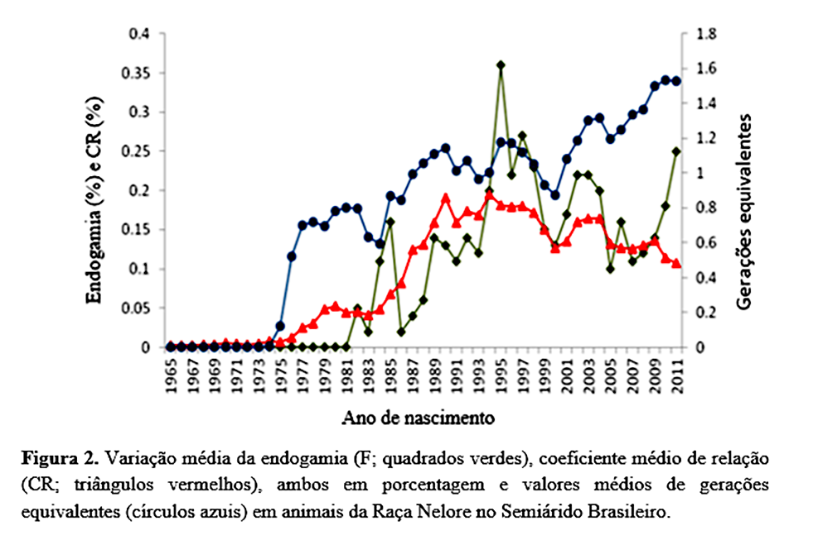
O mesmo comportamento pode ser observado entre 1996 a 2011, quando ocorreu um incremento da endogamia (0,25% - 1,53 gerações equivalentes). Percebe-se que a flutuação dos valores de endogamia segue os valores das gerações equivalentes, uma vez que, quanto mais se conhece as gerações que separam o indivíduo de cada ancestral conhecido, melhor a estimativa da endogamia.
De acordo com Queiroz et al.(6), o efeito da endogamia acarreta a elevação da frequência de genótipos envolvendo genes recessivos, comumente encobertos pelos alelos dominantes. Estes alelos recessivos podem tanto gerar disfunções e má formação nos animais, como interferir no desempenho das características de importância econômica. Vasconcelos et al.(29) reforçam a perda de variabilidade genética e consequente diminuição das oportunidades para realização da seleção e melhoramento como aspecto importante gerado pela elevação da endogamia.
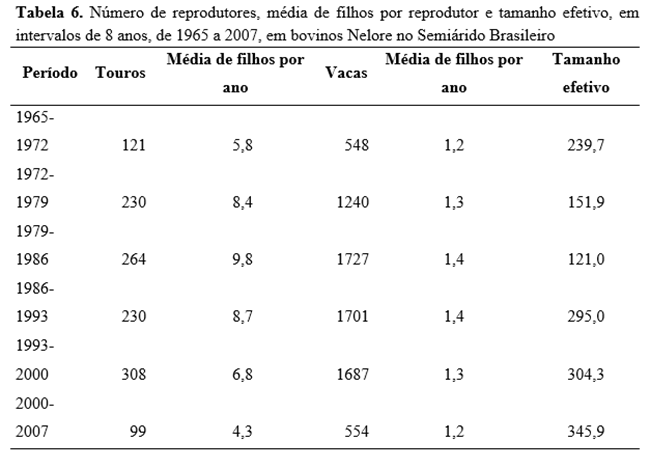
Após o ano de 1994, o coeficiente de relação médio (CR) passou por oscilações que acompanhavam as gerações equivalentes, atingindo seu ponto máximo em 0,20% neste ano (1,0 geração equivalente). O Ne pode apresentar valores maiores ou menores conforme a utilização de reprodutores no rebanho, ou seja, a sua redução pode se dar a partir da intensificação do uso de alguns reprodutores no rebanho associada, assim, ao aumento da progênie por reprodutor. Durante os anos de 1965-2007, obtiveram-se 36 períodos considerando-se intervalos de 8 anos, em que os Ne variaram de 74,9 a 607,3, entre 1974-81 e entre 1998-2005, respectivamente.
Vale ressaltar que Ne da presente pesquisa foi calculado pela variância da família e, de acordo com Gutierrez et al.(30), o Ne computado a partir de variâncias de tamanho das famílias não é útil para caracterizar o “real effective size”. No entanto, esses autores afirmaram que Ne calculado desta forma reflete uma política de acasalamento temporária e pode ser útil quando o conhecimento do pedigree é limitado e/ou a subdivisão ainda não ocorreu.
Durante os anos avaliados, foi notório o decréscimo na proporção fêmea:macho a partir de 1992, sendo acentuada em 1998 a diminuição da utilização de fêmeas e machos, semelhante aos valores encontrados anteriormente a 1974, bem como o número de filhos por reprodutor (4,3; 4,1 e 4,3) nos três últimos períodos (Tabela 6).
O rebanho Nelore criado no Semiárido Nordestino apresentou intervalo médio de gerações de 8,3 ± 4,1 anos, em que os menores valores foram obtidos entre os intervalos mãe-filho e mãe-filha (7,5 ± 3,3 e 7,7 ± 3,5) (Tabela 7). Para as estimativas do intervalo de gerações, Malhado et al.(31) verificaram nos rebanhos Nelore no estado da Bahia valores de 9,1±4,6 (pai-filho), 9,0±4,5 (pai-filha), 7,6±3,6 (mãe-filho), 7,5±3,5 anos (mãe-filha).
Otimizar o intervalo médio de gerações é essencial em programas de melhoramento genético, considerando que intervalos grandes diminuem o ganho genético anual quanto às características selecionadas, o que leva a menor retorno econômico do programa. Malhado et al.(31) reforçam, que para elevar os ganhos genéticos, é fundamental a diminuição do intervalo de geração, o aumento do tamanho efetivo, o uso de reprodutores avaliados e o controle dos acasalamentos de animais aparentados.
Prolongar a utilização de reprodutores permite que animais com idades avançadas sejam aproveitados por longos períodos. Esse processo é demonstrado em todas as passagens gaméticas, principalmente na pai-filho e pai-filha (8,7±4,2, em ambos), que obtiveram os maiores valores.
Conforme estudo de Faria et al.(32), a utilização de touros por longos períodos na reprodução e em centrais de inseminação contribuem na elevação dos níveis de endogamia e acarretam a redução do tamanho efetivo da população de bovinos Nelore, parâmetros essenciais para o monitoramento da variabilidade genética ao longo dos ciclos seletivos.
Falcão et al.(25) enfatizam que, apesar de apresentar ganho na produtividade do rebanho, é preocupante o uso extensivo de poucos touros, pois isto pode levar à redução da variabilidade genética e a efeitos deletérios decorrentes da endogamia. Portanto, sugere-se uma cautela ao considerar a longevidade reprodutiva no sistema de produção, pois quando há ausência de uma estratégia de acasalamento criteriosa, há um risco de ocorrerem cruzamentos consanguíneos, o que pode gerar um incremento endogâmico e uma diminuição do Ne e do ganho genético.
Conclusão
O rebanho possui número reduzido de animais na formação genética e pequena integralidade do pedigree, dificultando a estimativa de alguns parâmetros populacionais. Recomenda-se a diminuição do intervalo de geração. O menor número de registros de machos nos primeiros anos pode ter contribuído para o acasalamento de animais aparentados.
Referências
1. Souza GS. Previsões para o mercado de carnes. Revista de Economia e Sociologia Rural. 2011:49:(2):473-492.
2. Boligon AA, Albuquerque LG, Rorato PRNR. Associações genéticas entre pesos e características reprodutivas em rebanhos da raça Nelore. Revista Brasileira de Zootecnia. 2008:37:(4): 596-601.
3. Correa MBB, Dionello NJL, Cardoso FF. Genotype environment interaction effect on beef cattle genetic evaluation. Revista Brasileira de Agrociência. 2007:13:(2):153-159.
4. Carneiro PLS, Malhado CHM, Euclydes RF, Carneiro APS, Cunha EE. Endogamia, fixação de alelos e limite de seleção em populações selecionadas por métodos tradicionais e associados a marcadores moleculares. Revista Brasileira de Zootecnia. 2007:36:(2): 369-375.
5. Malhado CHM, Carneiro PLS, Martins Filho R, Azevedo DMMR. Histórico genético e populacional do rebanho Nelore Puro de Origem no Sertão Nordestino. Pesquisa Agropecuária Brasileira. 2009:44:(7):713-718.
6. Queiroz AS, Albuquqerque LG, Lanzoni NA. Efeito da endogamia sobre características de crescimento de bovinos da raça Gir no Brasil. Revista Brasileira de Zootecnia. 2000:29:(4):1014-1019.
7. Maignel L, Boichard D, Verrier E. Genetic variability of French dairy breeds estimated from pedigree information. Interbull Bull. 1996:14:(1): 49-54.
8. Meuwissen TI, Luo Z. Computing inbreeding coefficients in large populations. Genetics Selection Evolution. 1992:24:(1):305-313.
9. Boichard D, Maignel L, Verrier E. The value of using probabilities of gene origin to measure genetic variability in a population. Genetics Selection Evolution. 1997:29:(1): 5-23.
10. Lima RCC, Cavalcante AMB, Perez-Marin AM. Desertificação e mudanças climáticas no semiárido brasileiro, Campina Grande: INSA-PB, 2011.
11. INSA, Instituto Nacional do Semiárido. Pesquisas e projetos. Disponível em: <http://www.insa.gov.br/?page_id=112>, Acesso em: 08 de abril de 2015.
12. Gutiérrez JP, Goyache F. A Note on ENDOG a computer program for analysing pedigree information. Journal of Animal Breeding and Genetics. 2005:122:(3):172-176.
13. Maccluer J, Boyce AJ, Dyke B, Weitkamp LR, Pfenning QW, Parsons CJ. Inbreeding and pedigree structure in Standardbred horses. Journal of Heredity. 1983:74:(6):394-399.
14. Hill WG. A note on effective population size with overlapping generations. Genetics. 1979:92:(1):317-322.
15. Quaas RL. Computing the diagonal elements of a large numerator relationship matrix. Biometrics. 1976:32:(1): 949-953.
16. James JWA note on selection differentials and generation length when generations overlap. Animal Production. 1977:24:(1):109-112.
17. Cunha EE, Euclydes RF, Torres RA, Carneiro PLS. Simulação de dados para avaliação genética de rebanhos de gado de corte. Arquivo Brasileiro de Medicina Veterinária e Zootecnia. 2006:58:(3):381-387.
18. Barros EA, Ribeiro MN, Almeida MJO, Araujo AM. Population structure and genetic variability of the Marota Goat breed. Archivos de Zootecnia. 2011:60:(231): 543-552.
19. Vozzi PA, Marcondes CR, Magnabosco CU, Bezerra LAF, Lobo RB. Structure and genetic variability in Nellore (Bos indicus) cattle by pedigree analysis. Genetics and Molecular Biology. 2006:29:(3):482-485.
20. Muniz LMS, Souza LA, Barbosa ACB, Ambrosini DP, Oliveira AP, Carneiro PLS, Malhado CHM, Martins Filho R, Duarte RAB. The Polled Gir Polled in Northeastern Brazil: population genetic structure via analysis of pedigree. Arquivos Brasileiros de Medicina Veterinária e Zootecnia. 2012:64:(6):1656-1664.
21. Canaza-Cayo AW, Lopes OS, Silva MVGB, Cobuci JÁ, Torres RA, Martins MF, Arbex, WA. Population structure of Girolando breed. Ciência Rural. 2014:44:(11): 2072-2077.
22. Carneiro PLS, Malhado CHM, Martins Filho R, Carneiro APS, Silva FF, Torres RA. A raça Indubrasil no Nordeste brasileiro: melhoramento e estrutura populacional. Revista Brasileira de Zootecnia. 2009:38:(12): 2327-2334.
23. Faria FJC, Vercesi Filho AE, Madalena FE, Josahkian LA. Genetic structure of the registred polled Gir breed. Boletim da Industria Animal. 2006:63:(3):135-141.
24. ABCZ. Associação Brasileira dos Criadores de Zebu. Estatísticas. Disponível em: <www.abcz.org.br/ conteudo/tecnica/estatisticas.html> Acesso em: 15 junho de 2013.
25. Falcão AJS, Martins Filho R, Mabnabosco CU, Bozzi R, Lima FAM. Effects of Inbreeding on Reproductive and Growth Traits, and Breeding Values in a Closed Brown Swiss Herd. Revista Brasileira de Zootecnia. 2001:30:(1):83-92.
26. Carvaleiro R, Pimentel ECG. Endogamia: possíveis consequências e formas de controle em programas de melhoramento de bovinos de corte. Workshop em genética e melhoramento na pecuária de corte. 2004:2:10.
27. Gutiérrez JP, Altarriba J, Diaz C, Quintanilla R, Canon J, Piedrafita J. Pedigree analysis of eight Spanish beef cattle breeds. Genetics Selection Evolution. 2003:35:(1):43-63.
28. Goyache F, Fernandez I, Espinosa MA, Payeras L, Perez-Pardal L, Gutiérrez JP, Royo LJ, Alvarez I. Demographic and genetic analysis of the Mallorquina sheep flockbook. Itea. 2010:106:(1):3-14.
29. Vasconcelos J. Inbreeding and inbreeding depression in portuguese dairy cattle. Revista Portugual de Ciências Veterinarias. 2005:100:(553-554):33-38.
30. Gutiérrez JP, Cervantes I, Molina A, Valera M, Goyache F. Individual increase in inbreeding allows estimating effective sizes from pedigrees. Genetics Selection Evolution. 2008:40:(4):359-378.
31. Malhado CHM, Carneiro PLS, Pereira DG, Martins Filho R. Progresso genético e estrutura populacional do rebanho Nelore no Estado da Bahia. Pesquisa Agropecuária Brasileira. 2008:43:(9):1163-1169.
32. Faria FJC, Vercesi Filho AE, Madalena FE, Josahkian LA. Pedigree analysis in the Brazilian Zebu. Journal Animal Breed Genetics. 2009:126:(2):148-153.