

Resumo
A natureza ubíqua dos enterococos e sua capacidade de colonizar diferentes habitats são responsáveis pela sua fácil disseminação pela cadeia alimentar. No presente estudo, avaliamos a distribuição e a susceptibilidade antimicrobiana de isolados de Enterococcus provenientes de produtos cárneos. Cem produtos (carne de frango cru, carne de porco crua e carne cozida) foram adquiridos e cultivados para a presença de Enterococcus spp. No total, 194 amostras foram avaliadas, com taxas de contaminação de 63,6% nas amostras de frango, 31% na carne de porco crua e 1,4% nas amostras de carne cozida. A amplificação por PCR foi realizada para confirmar a presença de Enterococcus spp. (95/96), E. faecalis (66/96), E. faecium (30/96) E. casseliflavus/E. flavescens (3/96). Resultados de susceptibilidade mostraram que 100% dos isolados foram resistentes a pelo menos um antibiótico, sendo 100% de E. faecium resistentes a vancomicina, estreptomicina, ciprofloxacina, norfloxacina, eritromicina e tetraciclina. E. casseliflavus / E. flavescens resistentes a gentamicina, estreptomicina, ciprofloxacina, norfloxacina, eritromicina e tetraciclina. E. faecalis foram resistentes a ciprofloxacina, tetraciclina e eritromicina (92%), norfloxacina (83%), vancomicina e estreptomicina (50%). Na genotipagem, foram detectados os genes tetL e vanB. A presença desses microrganismos resistentes aos antimicrobianos nos alimentos pode causar problemas para a saúde pública.
Palavras-chaves: Antibióticos, enterococci vancomicina-resistante, PCR.
Abstract
The ubiquitous nature of enterococci and their ability to colonize different habitats account for their easy spread throughout the food chain. Here, we evaluated the distribution and antimicrobial susceptibility of Enterococcus isolates from meats obtained from different supermarkets. We acquired and cultured 100 products (raw chicken meat, raw pork, and boiled meats) to screen for the presence of Enterococcus spp. In total, 194 isolates were recovered from the samples, with contamination rates of 63.6% in the chicken samples, 31% in the raw pork meat, and 1.4% in the boiled meat samples. PCR amplification with specific primers was performed to screen the DNA of Enterococcus spp. (95/96), E. faecalis (66/96), E. faecium (30/96), and E. casseliflavus/E. flavescens (3/96). The antimicrobial susceptibility tests showed that all the isolates were resistant to at least one of the antibiotics. All E. faecium isolates were resistant to vancomycin, streptomycin, ciprofloxacin, norfloxacin, erythromycin, and tetracycline. The E. casseliflavus/E. flavescens isolates were resistant to gentamicin, streptomycin, ciprofloxacin, norfloxacin, erythromycin, and tetracycline. E. faecalis isolates were resistant to ciprofloxacin, tetracycline, and erythromycin (92%), norfloxacin (83%), vancomycin, and streptomycin (50%). The resistance genes tetL and vanB were detected by genotyping. The presence of these antimicrobial-resistant microorganisms in food might pose problems for public health.
Keywords: Antimicrobials, PCR, vancomycin-resistant enterococci
Seção: Ciência e Tecnologia de Alimentos
Recebido
14 de março de 2019.
Aceito
30 de outubro de 2019.
Publicado
22 de abril de 2020.
www.revistas.ufg.br/vet
Como citar - disponível no site, na página do artigo.
Introdução
Os enterococos são bactérias comensais do intestino de humanos e animais e causam doenças em pacientes imunocomprometidos, internados em unidades de terapia intensiva por longos períodos ou com doenças subjacentes graves(1); podem sobreviver em uma variedade de ambientes, como solo, água, plantas e alimentos(2,3). Os enterococos são contaminantes frequentes da carne de aves e, também, são agentes bem conhecidos na cadeia alimentar(4). Embora o significado dos enterococos resistentes a antimicrobianos nos alimentos não tenha sido bem elucidado, as bactérias que apresentam características relacionadas à resistência no ambiente podem ser consideradas reservatórios genéticos desses genes(5).
Os enterococos têm altos níveis de resistência a classes de antibióticos medicamente importantes, particularmente àquelas aprovadas pelo FDA para uso na produção animal, incluindo macrólidos, cloranfenicol, aminoglicosídeos e vancomicina(6,7). A vancomicina é um agente importante para o tratamento de infecções graves causadas por espécies enterocócicas; no entanto, a eficácia terapêutica desse antibiótico foi limitada pelo surgimento de enterococos resistentes à vancomicina (ERV)(8). O gene de resistência à vancomicina mais importante é o vanA. Várias espécies de enterococos, incluindo E. durans, E. hirae, E. gallinarum, E. casseliflavus, E. raffinosus, E. avium e E. mundtii têm esse gene(9).
Enterococos resistentes a antibióticos, isolados de animais e alimentos foram descritos na Europa e nos EUA(10,11). No Brasil, essas bactérias foram relatadas nos estados do Sul(12,13). Anteriormente, pesquisadores(14) relataram a presença de espécies de Enterococcus resistentes a antimicrobianos em queijo na região Sul do Brasil, embora os dados sobre a ocorrência de genes de resistência e resistência em enterococos do Paraná sejam escassos. No entanto, a presença de enterococos nos alimentos é motivo de debate, pois algumas espécies de Enterococcus estão envolvidas em infecções clínicas, como endocardite, bacteremia, infecções do trato urinário e sepse neonatal(3). Embora cepas de ERV isoladas de animais tenham sido estudadas extensivamente, pouco se sabe sobre a resistência combinada nesses microrganismos.
Assim, espera-se que nosso presente estudo sobre Enterococcus spp. associado a carnes cruas e cozidas forneça informações sobre a biodiversidade das espécies e a potencial resistência dessas bactérias, que são de extrema importância para garantir a segurança alimentar e a saúde pública.
Materiais e métodos
Um total de 100 amostras de alimentos foram obtidas de diferentes supermercados, sendo: 66 amostras de carne de frango crua, 14 amostras de carne de porco crua, 10 amostras de produtos processados à base de carne de porco (carne de porco picada, presunto, salsicha, mortadela e linguiças cozidas e não cozidas) e 10 produtos à base de carne de frango (linguiça, creme, frango à milanesa e besteira). Resumidamente, as amostras de carnes (25 g) foram coletadas assepticamente, colocadas em 225 ml de caldo de infusão cerebral (BHI), homogeneizadas e incubadas por 24 horas a 37 °C. Após a pré-incubação, 0,1 mL do enriquecimento primário foi riscado em agar de canamicina esculina azida (KEA) e incubado por 24 horas a 37 °C. Colônias típicas, sugestivas de Enterococcus, foram selecionadas aleatoriamente em cada uma das culturas de isolamento primário nas placas KEA e submetidas a exame microscópico, coloração de Gram, teste de catalase e crescimento em caldo de BHI contendo NaCl a 6,5% a 10 °C e 45 °C(15).
O DNA genômico foi extraído pela técnica da fervura(14). A identificação de enterococos e a detecção de genes de resistência foram confirmadas pela Reação em Cadeia da Polimerase (PCR), utilizando iniciadores específicos e direcionados, previamente relatados(16) (Tabela 1). Todas as reações no termociclador (Bio-Rad Thermal Cycles T100TM) foram realizadas com um volume final de 20 μL e consistiam em 2 μL de DNA total (20 ng mL-1), 11,5 μL de água ultrapura, 2 μL de tampão Taq (10X ), 1 μL de MgCl2 (2,5 mM), 1,4 μL de dNTPs (0,17 mM), 1 μL de cada iniciador direto e reverso (20 μmol) (Tabela 1) e 0,1 μL de Taq DNA polimerase (1 U) (Invitrogen). O programa utilizado consistiu em 5 minutos de desnaturação a 95 °C , seguido de 30 ciclos de 30 s a 95 °C, 30 s a 56 °C, 30 s a 72 °C, e extensão final a 72 °C por 5 min. Os amplicons foram separados por eletroforese em géis de agarose a 1% contendo brometo de etídio, e a visualização por luz UV foi realizada por um sistema fotográfico computadorizado (BioDoc-it System, UVP). Um marcador de DNA de 100 pb (escada de DNA de 100 pb, New England Biolabs / EUA) foi usado para estimar o tamanho do produto amplificado.

Todos os isolados foram caracterizados por testes de suscetibilidade antimicrobiana com 12 antibióticos, utilizando o método de difusão em disco, de acordo com as diretrizes do Clinical and Laboratory Standards Institute(17). Os 12 antibióticos testados incluíram ampicilina (10 mg), ciprofloxacina (5 mg), cloranfenicol (30 mg), eritromicina (15 mg), estreptomicina (300 µg), gentamicina (120 µg), norfloxacina (10 µg), penicilina (10 μg), tetraciclina (30 mg), teicoplanina (30 mg) e vancomicina (30 mg). O antibiótico imipenem (100 mg) também foi testado. A concentração inibitória mínima (CIM) da vancomicina foi determinada pela técnica de microdiluição em caldo e os pontos de interrupção dos antibióticos utilizados foram os especificados pelas diretrizes do CLSI. Amostras de Staphylococcus aureus ATCC 25923 (American Type Culture Collection) foram usadas para controle de qualidade para os testes de suscetibilidade.
Resultados e discussão
Este estudo fornece a primeira descrição de enterococos resistentes a antimicrobianos isolados de carnes cruas e processadas do Paraná. Os enterococos constituem um grupo interessante de microrganismos porque são comensais de humanos e animais que ocorrem e crescem em uma variedade de alimentos e, também, foram implicados em infecções nosocomiais. Entre as 100 amostras de carne crua e processada coletadas de diferentes fontes de supermercados no Paraná, foram encontrados 194 isolados enterocócicos presuntivos em 96 amostras de alimentos, com base na coloração de Gram, ausência de atividade da catalase e crescimento em caldo de BHI com NaCl a 6,5% em temperaturas de 10 °C e 45 °C. Utilizando amplificação por PCR com iniciadores específicos, Enterococcus spp. (95/96), E. faecalis (66/96), E. faecium (30/96) e E. casseliflavus / E. flavescens (3/96) foram identificados com base no tamanho do amplicon obtido(14,18) (Figura 1).
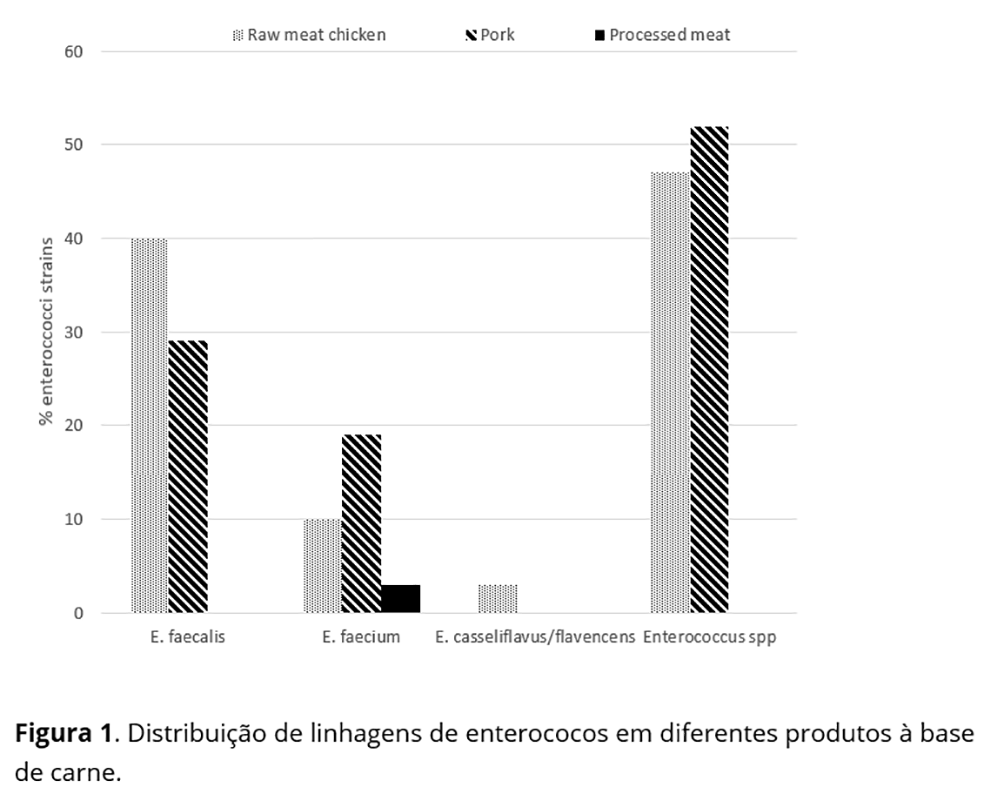
Em contraste com alguns relatos, E. casseliflavus / E. flavescens foram encontrados em nossas amostras de alimentos. Nossos resultados provavelmente representam a presença de uma etapa de enriquecimento. A prevalência relativa de espécies de Enterococcus em alimentos apresentou maior frequência para E. faecalis, seguida por E. faecium(4,12,19). Embora as espécies E. casseliflavus / E. flavescens não tenham sido isoladas com tanta frequência, em nosso estudo conseguimos isolar esse microrganismo, portanto essa espécie é importante porque tem resistência intrínseca à vancomicina(20). A presença da espécie E. flavescens não pôde ser confirmada entre as amostras analisadas.
Os dados aqui apresentados revelam novos caminhos para estudar um possível papel para a presença de Enterococcus spp. na cadeia alimentar, principalmente em carnes cruas e processadas. Portanto, é essencial investigar amostras adicionais de alimentos para determinar o risco potencial para a população.
A Tabela 2 mostra a resistência antimicrobiana e os genes de resistência presentes em cada cepa de Enterococcus spp. isolado de carnes cruas e processadas. Em comparação com outras espécies, a prevalência dos determinantes de resistência foi menor entre os isolados de E. faecium e E. casseliflavus / flavescens. Todos os isolados que apresentaram resistência à vancomicina foram submetidos a uma determinação da CIM. Como resultado, 100% dos isolados apresentaram resistência intermediária a esse antimicrobiano (CIM ≥ 8 µg / ml). Em relação à resistência antimicrobiana, os resultados do estudo mostraram que os isolados de Enterococcus spp., como E. faecalis e E. faecium, de amostras de carne de frango cru, foram mais frequentemente resistentes aos antimicrobianos testados do que os isolados de carne de porco crua e carne processada produtos.
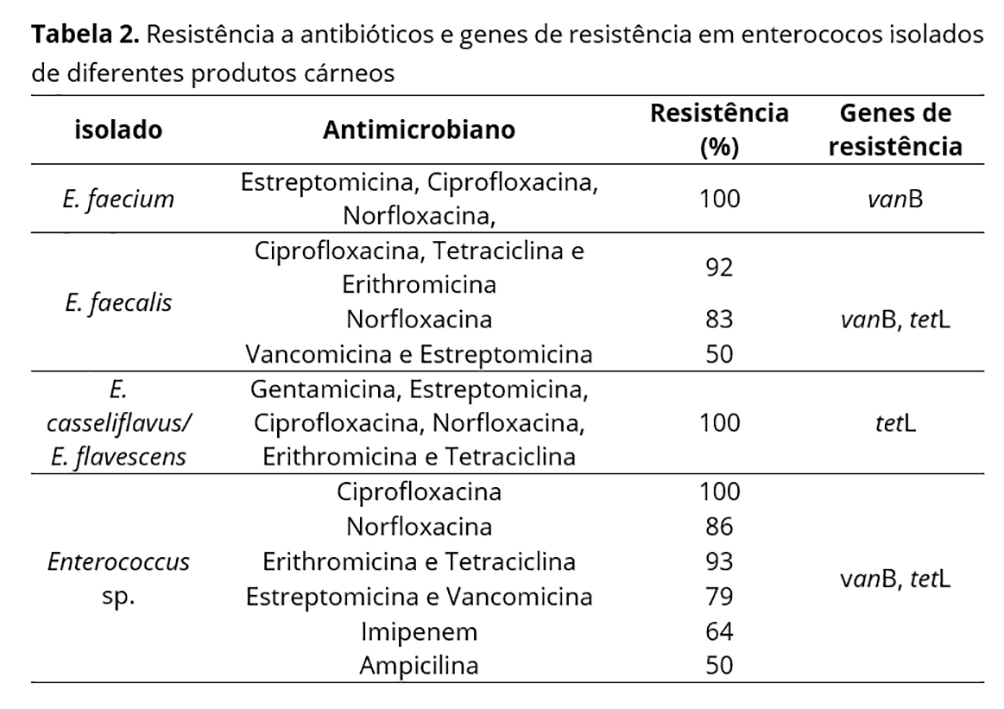
A distribuição de resistência a múltiplas drogas foi de 46%, 40%, 10% e 3,3% para Enterococcus spp., E. faecalis, E. faecium e E. casseliflavus / E. flavescens isolados, respectivamente. Todos os isolados de Enterococcus apresentaram diferentes perfis de resistência a múltiplas drogas, dos quais 35,7% dos isolados apresentaram resistência simultânea aos 12 antimicrobianos testados. Semelhante aos nossos resultados, esses autores(21) relataram que cepas de E. faecalis isoladas de alimentos eram mais resistentes a antibióticos do que outras espécies. Por outro lado, foi determinado que cepas de E. faecalis e E. faecium isoladas de alimentos prontos para consumo de origem animal apresentaram padrões semelhantes de resistência a antibióticos(22). Alguns pesquisadores relataram que cepas de E. faecium eram mais resistentes a antibióticos do que outras espécies(23,24).
As maiores taxas de resistência intermediária ao antimicrobiano cloranfenicol foram observadas para E. casseliflavus / E. flavescens (100%), seguidos por E. faecium (67%), E. faecalis (50%) e Enterococcus spp. (28%) Entre todas as amostras de E. faecalis, 33% apresentaram resistência intermediária à norfloxacina, seguida de 17% à ciprofloxacina e cloranfenicol. Entre os Enterococcus spp. isolados, foram demonstradas porcentagens mais altas de resistência intermediária para norfloxacina e tetraciclina (18%), seguidas de eritromicina e cloranfenicol (9%). Nenhum dos isolados de E. faecium apresentou resistência intermediária aos antimicrobianos testados.
Entre os isolados de E. faecalis, 17% foram resistentes à eritromicina e tetraciclina e 25% dos isolados de E. faecium apresentaram resistência à eritromicina. No outro, Enterococcus spp. isolados, não foi detectado perfil de resistência.
Nas amostras de produtos à base de carne processada, os isolados correspondentes às espécies de E. faecium foram identificados apenas nas amostras de carne de frango; esses isolados apresentaram perfil de sensibilidade para todos os antimicrobianos testados. Nenhum dos enterococos isolados da carne de porco crua ou processada mostrou um perfil de resistência a múltiplas drogas.
O perfil de suscetibilidade predominante entre os isolados de amostras de carne de frango crua foi o de resistência. Resultados semelhantes de resistência para isolados obtidos de alimentos foram descritos anteriormente(25). Relatos de resistência intermediária devem ser considerados um aviso, pois é provável que cepas intermediárias migrem para o grupo de cepas resistentes após o uso inadequado de antimicrobianos(13). Neste estudo, observamos que E. faecalis é mais intrinsecamente resistente a múltiplos agentes antimicrobianos, em particular a vários antibióticos β-lactâmicos e doses clínicas de aminoglicosídeos. No Brasil, não existem muitas informações sobre a resistência antimicrobiana de enterococos de alimentos(26).
Enterococcus spp. apresentaram resistência a entre 2 e 8 antibióticos. A resistência dos enterococos a múltiplos antibióticos não é surpreendente. Vários pesquisadores relataram que é comum os enterococos isolados de produtos fermentados de carne exibirem resistência a múltiplos antibióticos(27,28), como também confirmado neste estudo. Também foi demonstrado que cepas de Enterococcus isoladas de produtos fermentados à base de carne exibiram uma alta taxa de resistência a múltiplos antibióticos(27).
A resistência a antibióticos e a resistência a múltiplas drogas são problemas de saúde pública, pois podem causar a falha dos tratamentos para infecções enterocócicas. Embora as causas do desenvolvimento da resistência sejam contraditórias, evidências baseadas em dados de pesquisas sugerem uma ligação potencial entre a resistência antimicrobiana e as práticas veterinárias, como promoção do crescimento, profilaxia e tratamento(10,29,30).
Os dados sobre resistência a múltiplas drogas são extremamente preocupantes, principalmente devido ao risco de aquisição desses microrganismos e de seus elementos genéticos móveis, através da cadeia alimentar, do contato direto com animais ou mesmo da contaminação ambiental(4,31), que dificulta ainda mais o desenvolvimento de alternativas de tratamento para infecções humanas. Uma bactéria resistente pode infectar uma carcaça durante as operações de abate e resistir às barreiras existentes durante a produção de alimentos nas indústrias e/ou durante a preparação doméstica; então, pode atingir o trato gastrointestinal dos consumidores e modificar o perfil de resistência de sua microbiota intestinal(30).
No presente estudo, a base genotípica do fenótipo de resistência e/ou sensibilidade à tetraciclina e vancomicina encontrada nos isolados alimentares das cepas de Enterococcus foi investigada por PCR, com base na detecção dos genes tet e van. A distribuição do gene de resistência tetL, entre os isolados de alimentos, foi encontrada com maior frequência nos isolados de amostras de carne de frango cru. A presença do gene vanB foi evidente nos isolados de frango cru e carne de porco. O gene vanA não foi detectado em nenhum dos isolados estudados.
A frequência com que esses genes foram detectados neste estudo coincide com a da literatura(32;33). A fonte do ERV não é conhecida, embora os reservatórios em potencial desses organismos sejam hospitais e alimentos.
Cinco isolados de Enterococcus spp. não apresentaram os genes tetL, vanA ou vanB, e esses isolados ainda foram resistentes aos antimicrobianos testados, indicando que outros genes de resistência podem estar presentes.
A presença de um gene de resistência que não está sendo expresso fenotipicamente num determinado momento poderá estar em outro. Esses genes, chamados genes silenciosos, podem ser ativados por fatores ambientais, como as condições físicas do trato gastrointestinal ou os efeitos sinérgicos da microbiota bacteriana(34).
A resistência à vancomicina em enterococos isolados de alimentos tem um padrão variável no Brasil e em outros países, dependendo da região de origem(26). Deve-se considerar que a distribuição dos genes de resistência relatada neste estudo é baseada em um conjunto relativamente limitado de isolados, o que destaca a necessidade de monitoramento regular de diversas cepas para obter uma visão mais ampla da prevalência desses genes nos enterococos associados a alimentos.
Conclusão
Os enterococos são relativamente abundantes e resistentes às adversidades ambientais; portanto, têm sido propostos como indicadores bacterianos de resistência antimicrobiana, bem como de qualidade higiênica. No entanto, os enterococos emergiram como importantes patógenos oportunistas, com notável capacidade de expressar resistência a vários grupos de agentes antimicrobianos, limitando o número de opções terapêuticas. Utilizamos testes de difusão de disco com antibióticos e PCR para rastrear alguns genes de resistência de todos os isolados considerados neste estudo.
A transmissão de Enterococcus spp. pode ocorrer via alimento, porém ainda há poucas informações sobre a diversidade e distribuição de resistência nas espécies de Enterococcus isoladas da região norte do Paraná-Brasil. Este estudo fornece a primeira análise detalhada da resistência a antibióticos em uma variedade de enterococos isolados de carnes cruas e processadas nesse estado. Os enterococos devem ser considerados não apenas patógenos em potencial, mas também reservatórios de genes que codificam a resistência a antibióticos, que podem ser transferidos para outros microrganismos. O monitoramento contínuo de sua incidência e resistência emergente é importante para identificar alimentos que potencialmente representam um risco para a população e para garantir o tratamento eficaz de infecções enterocócicas humanas.
Agradecimentos
Os autores agradecem à UTFPR, ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) – Brasil e à Fundação Araucária – Paraná pelo apoio financeiro e recurso em bolsa de pós-graduação.
Referências
1.Foka F.E.T., Collins Njie Ateba. Detection of Virulence Genes in Multidrug Resistant Enterococci Isolated from Feedlots Dairy and Beef Cattle: Implications for Human Health and Food Safety. BioMed Research International. 2019; ID 5921840.
2. Gevers D, Danielsen M, Huys G, Swings J. Molecular characterization of tet(M) genes in Lactobacillus isolates from different types of fermented dry sausage. Applied. Environmental Microbiology. 2003; 69:1270–1275.
3. Donabedian SM. et al. Characterization of vancomycin-resistant Enterococcus faecium isolated from Swine in three Michigan counties. J Clin Microbiol. 2010; 48:4156-4160.
4. Bortolaia V., Espinosa-Gongora C., Guardabassi L.. Human health risks associated with antimicrobial-resistant enterococci and Staphylococcus aureus on poultry meat. Clin Microbiol Infect. 2016; 2:130-140.
5. Hayes JR. et al. Multiple-antibiotic resistance of Enterococcus spp. isolated from commercial poultry production environments. Appl Environ Microbiol, 2004; 70:6005-6011.
6. Manson AL, Van Tyne D, Straub TJ, Clock S, Crupain M, Rangan U, Gilmore MS, Earl A.M. Influence of agricultural antibiotic use on chicken meat-associated enterococci and their connection to the clinic. Appl Environ Microbiol. 2019; 30: AEM.01559-19.
7. Leavis HL, Willems RJ. Identification of high-risk enterococcal clonal complexes: global dispersion and antibiotic resistance. Curr Opin Microbiol. 2006; 9:454-460.
8. Sahlström L. Vancomycin resistant enterococci (VRE) in Swedish sewage sludge. Acta Vet Scan. 2009; 51:01-09.
9. Werner G. et al. Emergence and spread of vancomycin resistance among enterococci in Europe. Euro Surveill. 2008; 13:.1-11.
10. Gousia P. et al. Antimicrobial resistance of major foodborne pathogens from major meat products. Foodborne Pathog Dis. 2011; 8:27-38.
11. Rizzotti L., Rossi F., Torriani S. Biocide and antibiotic resistance of Enterococcus faecalis and Enterococcus faecium isolated from the swine meat chain. Food Microbiol. 2016; 60:160-164.
12. Camargo CH. et al. Prevalence and phenotypic characterization of Enterococcus spp. isolated from food in Brazil. Braz J Microbiol, 2014; 45:111-115.
13. Campos ACFB. et al. Resistência antimicrobiana em Enterococcus faecalis e Enterococcus faecium isolados de carcaças de frango. Pesqui Vet Bras, 2013; 33: 575-580
14. Furlaneto-Maia L. et al. Antimicrobial resistance in Enterococcus sp isolated from soft cheese in Southern Brazil. Adv Appl Microbiol 2014; 4:175-181.
15. Teixeira LM.; FACKLAM RR. (2015) Enterococcus. In PR Murray, EJ Baron, JH Jorgensen, MA Pfaller, RH Yolken (eds), Manual of Clinical Microbiology. 11 ed. Washington (DC): American Society for Microbiology, p.422-433. ISBN 978-1-55581-738-1
16. Dutka-Malen S. et al. Detection of glycopeptides resistance genotypes and identification to the species level of clinically relevant Enterococci by PCR. J Clin Microbiol. 1995; 33:.24-27.
17. CLSI. Performance Standards for Antimicrobial Susceptibility Testing. 28th ed. CLSI Supplement M100. Wayne, PA: Clinical and Laboratory Standards Institute; 2018.
18. Harwood VJ. et al. Vancomycin-Resistant Enterococcus spp. Isolated from Wastewater and Chicken Feces in the United States. Appl Environ Microbiol. 2001; 67:4930-4933.
19. Pesavento G. et al. Prevalence and antibiotic resistance of Enterococcus spp. isolated from retail cheese ready-to-eat salads, ham and raw meat. Food Microbiol. 2014; 41:1-7.
20. Igbinosa, E.O., Beshiru A. Antimicrobial Resistance, Virulence Determinants, and Biofilm Formation of Enterococcus Species From Ready-to-Eat Seafood. Front Microbiol. 2019 Apr 18;10:728.
21. Yogurtcu NN, Tuncer Y. Antibiotic susceptibility patterns of Enterococcus strains isolated from Turkish Tulum cheese. Int. J. Dairy Technol. 2013;66:236–242.
22. Chajecka-Wierzchowska W. et al. Occurrence and antibiotic resistance of enterococci in ready-to-eat food of animal origin. Afr. J. Microbiol. Res. 2012; 6:6773–6780.
23. Landeta G. et al. Technological and safety properties of lactic acid bacteria isolated from Spanish dry-cured sausages. Meat Sci. 2013; 95:272–280.
24. Valenzuela AS. et al. Virulence factors, antibiotic resistance and bacteriocins in enterococci from artisan food of animal origin. Food Control. 2009; 20:381–385.
25. Diarra MS. et al. Distribution of antimicrobial resistance and virulence genes in Enterococcus spp. and characterization of Isolates from broiler chickens. Appl Environ Microbiol. 2010; 76:8033-8043.
26. Cardoso, M. (2019), "Antimicrobial use, resistance and economic benefits and costs to livestock producers in Brazil", OECD Food, Agriculture and Fisheries Papers, No. 135, OECD Publishing, Paris.
27. Jahan M, Krause DO, Holley R A. Antimicrobial resistance of Enterococcus species from meat and fermented meat products isolated by a PCR-based rapid screening method. Int. J. Food Microbiol. 2013; 163:89–95.
28. Yüceer Ö, Özden Tuncer B. Determination of antibiotic resistance and biogenic amine production of lactic acid bacteria isolated from fermented Turkish sausage (sucuk) J. Food Safety. 2015;35:276–285.
29. Rosengren LB. et al. Associations between antimicrobial exposure and resistance in fecal Campylobacter spp. from grow-finish pigs on-farm in Alberta and Saskatchewan. J Food Prot. 2009;72:482-489.
30. Vignaroli C. et al. Multidrug-resistant enterococci in meat and faeces and co-transfer of resistance from an Enterococcus durans to a human Enterococcus faecium. Curr Microbiol. 2011; 62:1438-1447.
31. Aslam M. et al. Characterization of antimicrobial resistance and virulence genotypes of Enterococcus faecalis recovered from a pork processing plant. J Food Prot, 2012; 75: 1486-1491.
32. Terra M.R, Tosoni N.F, Furlaneto M.C, Furlaneto-Maia L. Assessment of vancomycin resistance transfer among enterococci of clinical importance in milk matrix, Journal of Environmental Science and Health, Part B. 2019.
33. Li S. et al. Vancomycin-resistant enterococci in a chinese hospital. Curr Microbiol, 2007; 55:125-127.
34. Chopra I, Roberts M. Tetracycline antibiotics: mode of action, applications, molecular biology, and epidemiology of bacterial resistance. Microbiol Mol Biol Rev. 2013; 65:.232-260.
35. Furlaneto-Maia L, Rocha KR, Siqueira VLD, Furlaneto MC. Comparison between automated system and PCR-based method for identification and antimicrobial susceptibility profile of clinical Enterococcus spp. Rev. Inst. Med. Trop. Sao Paul. 2014; 56(2):1-103. Disponível em https://www.researchgate.net/profile/Marcia_Furlaneto/publication/260809626_Comparison_between_automated_system_and_PCR-based_method_for_identification_and_antimicrobial_susceptibility_profile_of_clinical_Enterococcus_spp/links/53f47e0d0cf2fceacc6e83d1/Comparison-between-automated-system-and-PCR-based-method-for-identification-and-antimicrobial-susceptibility-profile-of-clinical-Enterococcus-spp.pdf